





Metodiche innovative di biologia molecolare per la genotipizzazione di virus, batteri, parassiti e l’individuazione di farmaco resistenza
Durata del progetto: 23 mesi
Investimento: 143.616,00
Contributo: 71.808,00
DESCRIZIONE DEL PROGETTO
Questo progetto nasce dalla necessità di sviluppare dispositivi diagnostici innovativi basati sulla tecnologia del Next Generation Sequencing (o NGS) in grado di reperire informazioni precise e accurate sui patogeni, in modo da orientare il clinico verso l’opzione terapeutica più adeguata, risparmiando tempo ed evitando la somministrazione di farmaci inadeguati che rischiano di far insorgere ulteriori farmacoresistenze. Per questa ragione si è deciso di concentrarsi su due target per cui ci sono necessità diagnostiche non soddisfatte.
Il primo ambito selezionato per lo sviluppo di un diagnostico è il virus dell’Epatite C (HCV), il dispositivo sviluppato prevede l’identificazione del virus dell’HCV e la sua genotipizzazione fornendo ai clinici importanti indicazioni riguardo la terapia più adatta da somministrare al paziente.
Il secondo diagnostico è improntato sull’identificazione e sulla determinazione delle resistenze antibiotiche dei patogeni causanti la sepsi, un’infezione sistemica altamente rischiosa per la vita del paziente.
Per far fronte a queste necessità è stata selezionata la tecnologia del Next Generation Sequencing (NGS) che permette la raccolta di un gran numero di dati genici dei patogeni in parallelo, consentendo l’ottenimento di preziose informazioni caratterizzanti il microrganismo. L’alta sensibilità di questa tecnica la rende adatta anche all’individuazione delle sottopopolazioni di microrganismi resistenti ai trattamenti all’interno di un insieme di ceppi sensibili.
OBIETTIVI
Il progetto ha come obiettivo la realizzazione di due differenti dispositivi diagnostici in vitro per l’identificazione e la determinazione delle resistenze dell’HCV e dei patogeni batterici e fungini coinvolti nell’insorgenza della sepsi. Per realizzare questi dispositivi è stata selezionata una tecnologia altamente innovativa, il sequenziamento massivo di nuova generazione (NGS).
RISULTATI
Durante il progetto sono state fatte ricerche sullo stato dell’arte identificando le carenze nei dispositivi diagnostici attualmente utilizzati in ambito ospedaliero. Per quanto riguarda l’HCV, è stata evidenziata l’assenza di diagnostici in grado di determinare il genotipo del virus con una metodologia affidabile e flessibile. L’importanza della genotipizzazione del virus risiede nella diversa suscettibilità dei vari genotipi ai trattamenti farmacologici; la determinazione del genotipo consentirà di indirizzare il clinico verso la terapia più efficace risparmiando tempo e migliorando la qualità della vita del paziente.
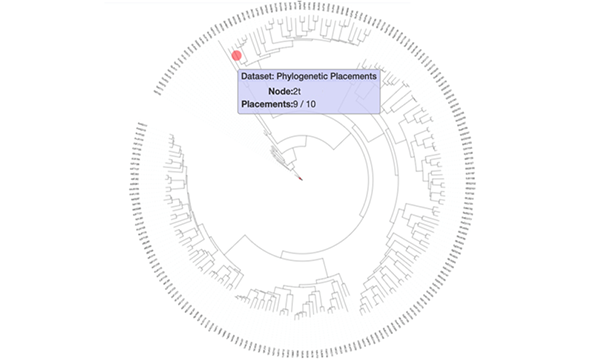
“Figura 2: Albero filogenetico del virus HCV
con identificazione del genotipo del campione”
È stato quindi messo a punto un kit diagnostico in grado di identificare il virus dell’epatite C e di determinarne il genotipo, lo sviluppo di questo diagnostico ha richiesto un’attenta progettazione dei primer necessari per l’amplificazione delle regioni geniche di interesse e un approfondito studio della filogenetica del virus. Il kit è composto dai reagenti da utilizzare per il sequenziamento del genoma virale e il software per l’interpretazione dei dati sviluppato in house.
Nel caso della sepsi, un’infezione sistemica potenzialmente letale, la rapidità nella diagnosi e nell’identificazione del patogeno coinvolto è essenziale, in quanto si è visto che l’aspettativa di vita del paziente diminuisce dell’8% per ogni ora di ritardo nella somministrazione della terapia. Attualmente i protocolli diagnostici si basano sulla microbiologia classica con un tempo di refertazione che supera le 24h. In questo progetto è stato sviluppato un kit innovativo per l’identificazione di un ampio pannello di patogeni coinvolti nella sepsi e per la determinazione dei geni e delle mutazioni geniche da essi veicolate con un Turn Around Time di meno di 12h.
Questa rapidità nell’ottenimento del risultato è stata ottenuta sviluppando il kit in modo da consentire l’identificazione dei patogeni da sangue, bypassando l’emocoltura, e selezionando un protocollo di preparazione delle librerie NGS di 3h.